Corynebacterium uropygiale ist ein Aktinobakterium, der Gattung Corynebacterium, welches bislang ausschließlich in Bürzeldrüsen (lat. Glandula uropygialis) von Truthühnern (Meleagris gallopavo) nachgewiesen wurde, wo es zum natürlichen Mikrobiom gehört. Obwohl Bakterien – darunter auch Corynebakterien – häufig auf Vögeln zu finden sind, ist C. uropygiale Seit seiner Neubeschreibung im Jahre 2016 durch Braun et al. der bisher einzige Vertreter seiner Gattung, dessen Lebensraum die Bürzeldrüse darstellt.
Allgemeine Eigenschaften
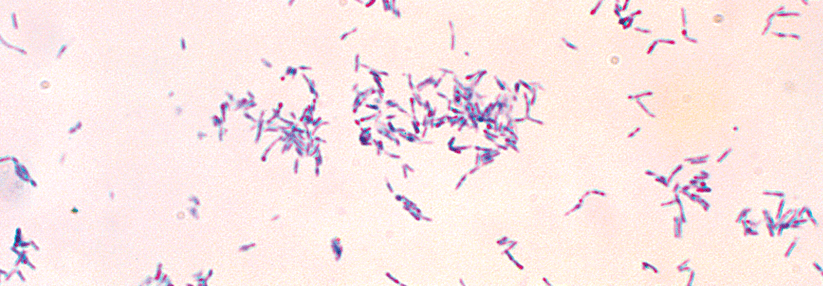
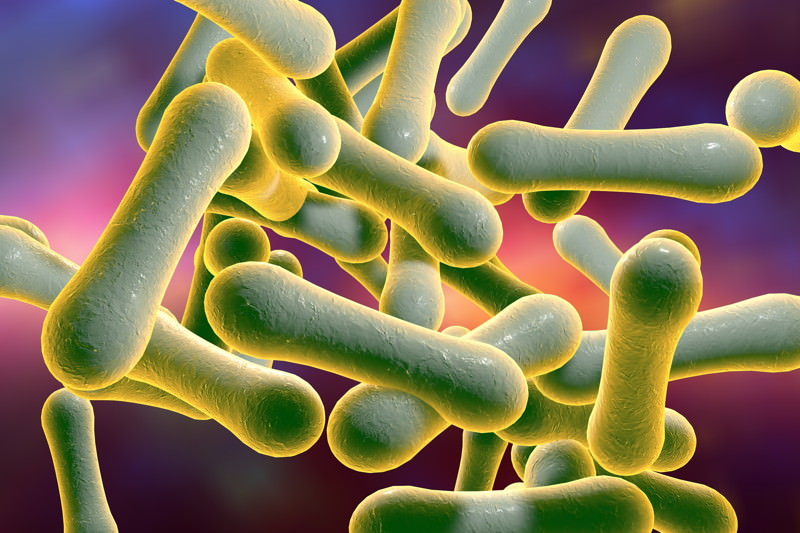
Corynebacterium uropygiale ist ein keulenförmiges Gram-positives Stäbchen, das häufig in Palisaden- bzw. V-Formationen angeordnet ist. Es handelt sich um nicht-säurefeste, nicht-sporenbildende fakultative Anaerobier. Wie bei den meisten Corynebacterien, enthält die Zellwand von C. uropygiale Mykolsäuren. Der G C-Gehalt des Typenstamms beträgt 60,7 mol% in the. Der nächste Verwandte der Art stammt wurde ebenfalls auf Individuen der Klasse der Vögel gefunden: 2003 wurde Corynebacterium spheniscorum durch Goyache et al. (2003) von Magellanpinguinen (Spheniscus magellanicus) isoliert.
Anzucht
Corynebacterium uropygiale wächst auf Komplexmedien, die einfach ungesättigte Fettsäuren, wie z. B. Tween-80 enthalten. Auf solchen Agarmedien ist nach 48 h Inkubation bei 37 °C gutes Wachstum zu verzeichnen. Auf Columbia-Agar mit 5 % Schafsblut wächst das Bakterium allerdings nur schlecht. Auf Medien wie Lysogeny Broth, Tryptic-Soy-Agar oder Müller-Hinton-Agar wächst C. uropygiale nicht.
Lipophilie
C. uropygiale vermag es, sich in lipidreicher Umgebung wie der Bürzeldrüse zu vermehren. Statt die dafür notwendigen Fettsäuren unter Energieaufwendung selbst zu produzieren, nutzt es Substanzen aus dem Bürzeldrüsensekret. Dies kann als evolutionäre Anpassung an das Habitat angesehen werden, denn die dadurch eingesparte Energie, kann nun anderen Stoffwechselvorgängen zugeführt werden, wodurch eine potentielle Steigerung der biologischen Fitness resultiert.
Weblinks
- Type strain of Corynebacterium uropygiale at BacDive - the Bacterial Diversity Metadatabase
Quellen